













中国农业科学 ›› 2025, Vol. 58 ›› Issue (11): 2045-2061.doi: 10.3864/j.issn.0578-1752.2025.11.001
• 作物遗传育种·种质资源·分子遗传学 • 下一篇
王晖1( ), 丁保朋2(
), 丁保朋2( ), 李彧贤2, 任泉如2, 周海1, 赵均良1,3(
), 李彧贤2, 任泉如2, 周海1, 赵均良1,3( ), 胡海飞3(
), 胡海飞3( )
)
收稿日期:2024-11-18
接受日期:2025-01-16
出版日期:2025-06-01
发布日期:2025-06-09
通信作者:
联系方式:
王晖,E-mail:2224168009@qq.com。
基金资助:
WANG Hui1( ), DING BaoPeng2(
), DING BaoPeng2( ), LI YuXian2, REN QuanRu2, ZHOU Hai1, ZHAO JunLiang1,3(
), LI YuXian2, REN QuanRu2, ZHOU Hai1, ZHAO JunLiang1,3( ), HU HaiFei3(
), HU HaiFei3( )
)
Received:2024-11-18
Accepted:2025-01-16
Published:2025-06-01
Online:2025-06-09
摘要:
全球人口的持续增长和气候变化给粮食供给带来了严峻挑战,粮食安全问题因此愈发突出。为了满足不断增长的人口对粮食的需求,提升作物产量并增强其对环境的适应性已成为农业领域的重要目标。在此背景下,基因组学被视为加速作物育种进程的重要手段。通过深入挖掘和利用作物优异功能基因信息,不仅能有效提高作物产量,还能增强其抗逆性和适应性,为保障全球粮食安全和实现农业可持续发展提供有力支撑。然而,传统的单一参考基因组往往无法全面反映作物在驯化和改良过程中所累积的所有基因组变异,导致研究者对功能基因及其调控网络的认识存在局限。随着高通量测序技术的不断发展,基因组学研究开始迈入泛基因组学时代。通过整合多个高质量基因组,构建涵盖物种基因序列全集的泛基因组,能精准地鉴定包括单核苷酸多态性(SNPs)及结构变异(SVs)在内的多种遗传变异,全面地捕获物种在不同品种、亚种及野生亲缘种中广泛存在的遗传多样性,为系统挖掘优异功能基因提供更完善的分析框架。通过结合多组学数据(如转录组、蛋白质组、表观组等),泛基因组研究能在更精细的水平上挖掘优异功能基因,进而为分子育种提供更具针对性和准确性的基因靶标。同时,借助CRISPR-Cas9等基因编辑技术,可进一步对重要基因位点进行定向改造,剔除影响作物生长的不利性状或强化其对环境胁迫的抗性,从而为培育兼具高产、优质和抗逆特性的新一代作物品种奠定坚实基础。本文阐述了目前泛基因组的主要构建方法及展现形式的研究进展,并系统地梳理了作物泛基因组的发展及其在育种改良中的应用,深入探讨了泛基因组在未来作物育种中面临的挑战,对如何更好地应用泛基因组进行作物遗传改良进行了讨论,为未来精准分子育种改良提供了新的思路和策略。
王晖, 丁保朋, 李彧贤, 任泉如, 周海, 赵均良, 胡海飞. 作物泛基因组研究进展与展望[J]. 中国农业科学, 2025, 58(11): 2045-2061.
WANG Hui, DING BaoPeng, LI YuXian, REN QuanRu, ZHOU Hai, ZHAO JunLiang, HU HaiFei. Research Progress and Prospects on Crop Pan-Genomics[J]. Scientia Agricultura Sinica, 2025, 58(11): 2045-2061.
表1
基因组测序、组装技术的发展及应用"
| 技术类型 Technology type | 技术名称 Technology name | 时间 Time | 优势与劣势 Advantage and disadvantage | 应用实例 Application example |
|---|---|---|---|---|
| 第一代测序及组装 First generation sequencing and assembly | Sanger测序 Sanger sequencing | 1977 [ | 优势:具备高准确性,低错误率,测序结果易于分析和解释。 劣势:测序通量低导致测序效率低,难以满足大规模基因组测序的需求。 Advantages: It possesses high accuracy with a low error rate, making the sequencing results easy to analyze and interpret. Disadvantages: The low sequencing throughput results in inefficient sequencing, making it difficult to meet the demands of large-scale genomic sequencing. | 水稻基因组[ Rice genome, Sorghum genome, Maize genome |
| BACs克隆测序 BACs cloning and sequencing | 1992 [ | 优势:高分辨率,低嵌合率,高遗传稳定性,可容纳300 kb DNA片段,有助于获得高质量的基因组序列。 劣势:文库的构建和筛选过程相对复杂,BAC克隆的测序和组装成本较高。 Advantages: High resolution, low chimeric rate, good genetic stability, and the ability to accommodate up to 300 kb DNA fragments, contributing to the generation of high-quality genomic sequences. Disadvantages: The processes of library construction and screening are relatively complex, and the sequencing and assembly costs of BAC clones are high. | ||
| 第二代测序及组装 Next-generation sequencing and assembly | Illumina短读长NGS Illumina Short Read NGS | 2008 [ | 优势:低成本、高通量,高准确性。 劣势:读长较短,在复杂基因组及重复序列的组装时面临较大困难。 Advantages: Low cost, high throughput, and high accuracy. Disadvantages: Shorter read lengths pose challenges for the assembly of complex genomes and repetitive sequences. | 芥菜基因组[ Mustard genome, Lettuce genome, Rapeseed genome, Soybean genome |
| 第三代测序及组装 Third generation sequencing and assembly | PacBio测序 PacBio sequencing | 2023 [ | 优势:PacBio HiFi读长高达15—20 kb,具有高分辨率和精准性,从而提高基因组组装的连续性和完整性。 劣势:测序成本较高,在大规模项目中的应用可能受到限制。 Advantages: PacBio HiFi offers read lengths of up to 15-20 kb, high resolution, and accuracy, thereby enhancing the continuity and completeness of genome assembly. Disadvantages: The sequencing cost is relatively high, which may limit its application in large-scale sequencing projects. | 面包小麦基因组[ Bread wheat genome, Potato genome |
| Nanopore测序 Nanopore sequencing | 2021 [ | 优势:Ultra-long超长读长,最长读长可达1 Mb。适用于从头组装基因组和解析复杂的结构变异。 劣势:测序错误率相对较高,测序成本高。 Advantages: Ultra-long read lengths, with the longest reads reaching up to 1 Mb, making it suitable for de novo genome assembly and resolving complex structural variations. Disadvantages: Relatively high sequencing error rates and high sequencing costs. | 水稻基因组[ Rice genome, Pearl millet genome | |
| 混合组装 策略 Hybrid assembly strategy | 二代测序+三代测序混合组装 Hybrid assembly using second-generation and third-generation sequencing | 2012 [ | 优势:三代测序的长读长优势可以弥补二代测序在重复序列组装上的不足,生成更完整、更连续的基因组序列。二代测序可用于校正三代测序,从而提高测序准确性。 劣势:二代和三代测序数据在读长、错误模式和覆盖度等方面存在差异,整合这些数据进行组装需要复杂的算法和计算资源。 Advantages: The long read lengths of third-generation sequencing can compensate for the shortcomings of second-generation sequencing in assembling repetitive sequences, generating more complete and contiguous genome sequences. Second-generation sequencing can be used to correct third-generation sequencing, thereby improving its accuracy. Disadvantages: There are differences between second- and third- generation sequencing data in terms of read length, error patterns, and coverage. Integrating these data for assembly requires complex algorithms and computational resources. | 拟南芥基因组[ Arabidopsis genome, Potato genome |
| PacBio HiFi测序技术+Nanopore超长读长测序技术+HiC染色质构象捕获技术 PacBio HiFi+Nanopore Ultra-long+HiC | 2022 [ | 优势:综合了长读长、高准确性和三维基因组结构信息,可实现无间隙基因组组装。 劣势:成本较高,分析技术复杂性较高,需要较多的计算资源及相应的生物信息分析工具。 Advantages: Combining long read lengths, high accuracy, and three- dimensional genome structure information enables gapless genome assembly. Disadvantages: It is costly, involves complex analytical algorithm, and requires substantial computational resources as well as specialized bioinformatics analysis tools. | 玉米T2T(端粒到端粒)基因组[ Maize T2T (telomere-to- telomere) genome, Sorghum T2T genome |
表2
当今重要植物和作物泛基因组研究的概述"
| 泛基因组构建方法 Method for pangenome construction | 物种名 Species names | 品种个数 Number of varieties | 测序方法 Sequencing methods | 参考文献 References |
|---|---|---|---|---|
| 棉花Gossypium hirsutum | 344 | Illumina HiSeq PacBio | [ | |
| 从头组装、比较基因 组De novo assembly, Comparative genomics | 西瓜Citrullus lanatus | 547 | Illumina PacBio | [ |
| 西瓜Citrullus lanatus | 27 | Illumina HiSeq PacBio HiFi | [ | |
| 西瓜Citrullus lanatus | 400 | Illumina | [ | |
| 大麦Hordeum vulgare | 20 | Illumina HiSeq PacBio | [ | |
| 大麦Hordeum vulgare | 76 | Illumina NovaSeq PacBio HiFi | [ | |
| 瓠Lagenaria siceraria | 197 | Illumina NextSeq | [ | |
| 珍珠粟Pennisetum glaucum | 10 | Illumina Pacbio HiFi | [ | |
| 地钱Marchantia polymorpha | 133 | Illumina HiSeq PacBio HiFi | [ | |
| 亚洲栽培稻和普通野生稻 Oryza sativa and Oryza rufipogon | 111 | PacBio | [ | |
| 杨树Populus przewalskii Maxim | 19 | Illumina HiSeq Nanopore | [ | |
| 梨Pyrus spp. | 7 | Illumina HiSeq PacBio HiFi | [ | |
| 土豆Solanum tuberosum | 296 | Illumina HiSeq | [ | |
| 土豆Solanum tuberosum | 42 | PacBio HiFi | [ | |
| 玉米Zea mays | 14 | Illumina HiSeq PacBio | [ | |
| 玉米Zea mays | 26 | PacBio | [ | |
| 甘蓝型油菜Brassica napus | 8 | Illumina HiSeq PacBio | [ | |
| 从头组装、图形化泛基因组 De novo assembly, Graph-based pangenome | 甘蓝Brassica oleracea | 11 | PacBio HiFi | [ |
| 栽培和野生大豆 Glycine max and Glycine soja | 26 | Illumina HiSeq PacBio | [ | |
| 谷子Setaria italica | 110 | Illumina NovaSeq Pacbio | [ | |
| 番茄Solanum lycopersicum | 11 | Illumina PacBio | [ | |
| 黄瓜Cucumis sativus | 11 | PacBio | [ | |
| 番茄Solanum lycopersicum | 31 | PacBio HiFi | [ | |
| 亚洲栽培稻和非洲栽培稻 Oryza sativa and Oryza glaberrima | 33 | PacBio | [ | |
| 亚洲栽培稻、非洲栽培稻、普通野生稻和短舌野生稻 Oryza sativa, Oryza glaberrima, Oryza rufipogon and Oryza barthii | 251 | Nanopore | [ | |
| 柑橘Citrus reticulata | 12 | Illumina HiSeq PacBio | [ | |
| 鹰嘴豆Cicer arietinum | 8 | Illumina HiSeq | [ | |
| 葡萄Vitis vinifera | 9 | Illumina HiSeq PacBio | [ | |
| 迭代组装 Iterative assembly | 无油樟Amborella trichopoda | 11 | Illumina HiSeq | [ |
| 栽培和野生大豆 Glycine max and Glycine soja | 1110 | Illumina HiSeq | [ |
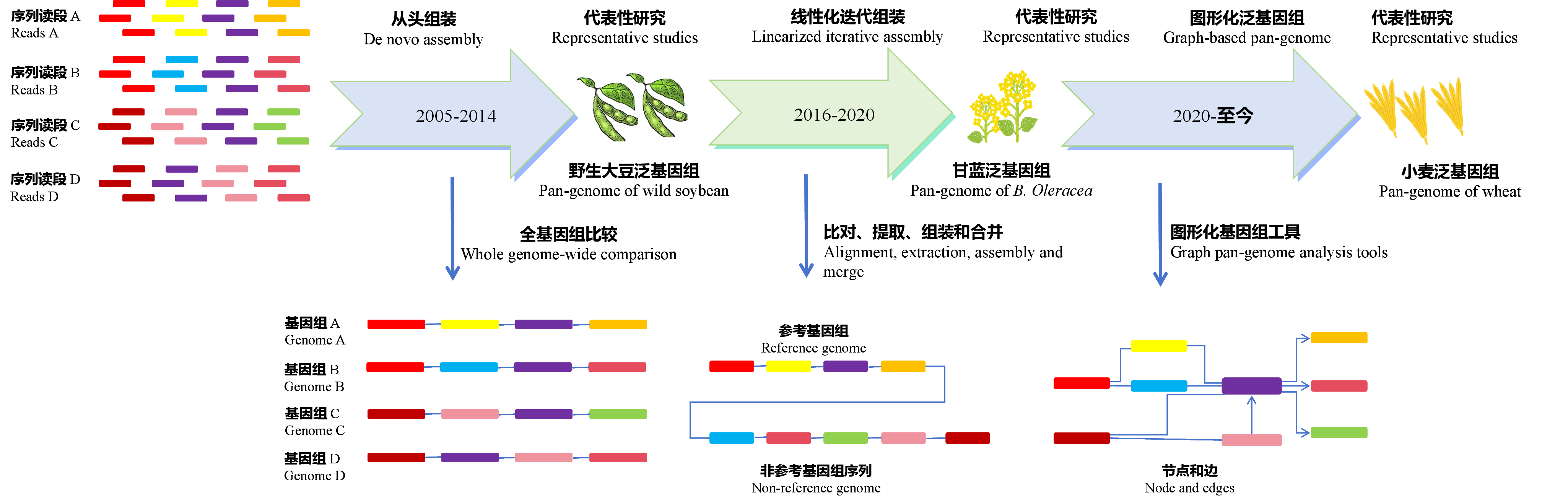
图1
泛基因组构建方法进展的时间线 2014年,第一个植物泛基因组[68](大豆)使用了de novo组装方法进行构建。该方法首先独立地组装每个基因组,随后对所有基因组序列进行比较,以识别核心基因组和非核心基因组。线性化迭代组装方法通过将reads(读长)与参考基因组(基因组A)比对,检测并组装未比对上的reads,最后将从新注释的新contigs添加到参考泛基因组中。此种方法首次应用于2016年构建的甘蓝[69]泛基因组,并在其他包含大量群体的泛基因组研究中得到了广泛的应用。图形化泛基因组在2020年首次开发,整合了泛基因组序列和变异信息。目前虽仍处于早期开发阶段,但已被运用于小麦等作物[70]"
| [1] |
|
| [2] |
doi: 10.1111/gcb.14381 pmid: 30084165 |
| [3] |
|
| [4] |
|
| [5] |
贺文闯, 许强, 钱前, 商连光. 水稻泛基因组学的发展与前景: 重要工具与应用. 生物技术通报, 2024, 40(10): 9-18.
doi: 10.13560/j.cnki.biotech.bull.1985.2024-0669 |
|
|
|
| [6] |
doi: 10.1038/s41477-020-0733-0 pmid: 32690893 |
| [7] |
刘羽诚, 申妍婷, 田志喜. 大豆泛基因组研究进展. 遗传, 2024, 46(3): 183-198.
|
|
|
|
| [8] |
doi: 10.1146/annurev-genet-021721-033821 pmid: 34310193 |
| [9] |
|
| [10] |
郝晨路, 於晓芬, 曲明昊, 赖恩惠, 郭素敏, 高磊. 植物泛基因组研究进展与展望. 植物科学学报, 2022, 40(1): 124-132.
|
|
|
|
| [11] |
|
| [12] |
doi: 10.1186/s13007-016-0107-9 pmid: 26793268 |
| [13] |
|
| [14] |
doi: 10.1126/science.1178534 pmid: 19965430 |
| [15] |
doi: 10.1146/annurev-genom-083115-022413 pmid: 27362342 |
| [16] |
|
| [17] |
|
| [18] |
|
| [19] |
|
| [20] |
|
| [21] |
doi: 10.1016/j.molp.2020.09.001 pmid: 32896642 |
| [22] |
|
| [23] |
doi: S0168-9525(18)30096-9 pmid: 29941292 |
| [24] |
doi: 10.1038/s41477-018-0289-4 pmid: 30390080 |
| [25] |
|
| [26] |
|
| [27] |
doi: 10.1038/s41588-022-01022-1 pmid: 35288708 |
| [28] |
|
| [29] |
|
| [30] |
|
| [31] |
|
| [32] |
doi: 10.1186/s13059-024-03252-4 pmid: 38671502 |
| [33] |
doi: 10.1186/s13059-020-1938-2 pmid: 32019604 |
| [34] |
|
| [35] |
doi: 10.1126/science.abj6987 pmid: 35357919 |
| [36] |
doi: 10.1038/s41588-023-01419-6 pmid: 37322109 |
| [37] |
|
| [38] |
|
| [39] |
doi: 10.1146/annurev.genom.9.081307.164359 pmid: 18576944 |
| [40] |
doi: 10.1038/s41592-022-01730-w pmid: 36635542 |
| [41] |
|
| [42] |
|
| [43] |
doi: 10.1038/s41588-020-0699-x pmid: 32989320 |
| [44] |
doi: 10.1016/j.cell.2021.04.046 pmid: 34051138 |
| [45] |
|
| [46] |
doi: 10.1038/s41422-022-00685-z pmid: 35821092 |
| [47] |
doi: 10.1101/gr.276015.121 pmid: 35396275 |
| [48] |
|
| [49] |
|
| [50] |
|
| [51] |
|
| [52] |
|
| [53] |
|
| [54] |
|
| [55] |
|
| [56] |
doi: 10.1186/s12915-024-01893-2 pmid: 38654264 |
| [57] |
|
| [58] |
|
| [59] |
|
| [60] |
|
| [61] |
doi: 10.1038/s41588-023-01302-4 pmid: 36864101 |
| [62] |
|
| [63] |
|
| [64] |
doi: 10.1038/s41467-022-28362-0 pmid: 35115520 |
| [65] |
|
| [66] |
|
| [67] |
|
| [68] |
|
| [69] |
|
| [70] |
|
| [71] |
|
| [72] |
|
| [73] |
doi: 10.1038/s41588-019-0410-2 pmid: 31086351 |
| [74] |
doi: 10.1093/plphys/kiad405 pmid: 37477947 |
| [75] |
|
| [76] |
|
| [77] |
doi: 10.1038/s41588-023-01340-y pmid: 37024581 |
| [78] |
|
| [79] |
|
| [80] |
doi: S0092-8674(20)30618-8 pmid: 32553274 |
| [81] |
|
| [82] |
|
| [83] |
doi: 10.1038/nbt.4227 pmid: 30125266 |
| [84] |
doi: 10.1186/s13059-020-02168-z pmid: 33066802 |
| [85] |
|
| [86] |
doi: 10.1038/s41592-024-02430-3 pmid: 39433878 |
| [87] |
王英豪, 余嘉鑫, 唐海宝, 张兴坦. 植物复杂基因组与泛基因组研究现状与展望. 中国科学: 生命科学, 2024, 54(2): 233-246.
|
|
|
|
| [88] |
|
| [89] |
|
| [90] |
|
| [91] |
doi: 10.1186/s13059-020-02224-8 pmid: 33397434 |
| [92] |
doi: S0092-8674(20)30616-4 pmid: 32553272 |
| [93] |
|
| [94] |
|
| [95] |
TAY FERNANDEZ C G,
|
| [96] |
|
| [97] |
|
| [98] |
|
| [99] |
|
| [100] |
doi: 10.1186/s13059-023-02861-9 pmid: 36703158 |
| [101] |
doi: 10.1038/s41592-022-01731-9 pmid: 36646895 |
| [102] |
|
| [103] |
doi: 10.1093/plcell/koae039 pmid: 38345423 |
| [104] |
|
| [105] |
doi: S1674-2052(16)30232-5 pmid: 27793787 |
| [106] |
doi: 10.1016/j.molp.2023.01.011 pmid: 36733253 |
| [1] | 唐桂梅, 李卫东, 周宇霞, 孔佑涵, 肖晓玲, 彭颖姝, 张力, 符红艳, 刘洋, 黄国林. 基于表型性状分析蕙兰种质资源遗传多样性[J]. 中国农业科学, 2025, 58(2): 339-354. |
| [2] | 李沛, 何治霖, 谈月霞, 赵婉彤, 冯锦英, 陈贵虎, 严池, 王子豪, 黄平, 江东. 基于重测序数据与表型性状的宽皮柑橘遗传多样性分析与优异种质筛选[J]. 中国农业科学, 2024, 57(23): 4761-4773. |
| [3] | 杨春, 杨代星, 李燕, 梁思慧, 邓小强, 乔大河, 陈娟, 郭燕, 林开勤, 陈正武. 贵州大树茶形态学特征及生化成分综合分析[J]. 中国农业科学, 2024, 57(19): 3894-3916. |
| [4] | 李玉姗, 肖菁, 马越, 田超, 赵连佳, 王帆, 宋羽, 蒋程瑶. 169份番茄种质资源表型性状遗传多样性分析及综合评价[J]. 中国农业科学, 2024, 57(18): 3671-3683. |
| [5] | 翟彩娇, 葛礼姣, 程玉静, 仇亮, 王小秋, 刘水东. 基于表型性状与SSR标记的冬瓜、节瓜种质资源遗传多样性分析[J]. 中国农业科学, 2024, 57(17): 3440-3457. |
| [6] | 雷梦林, 刘霞, 王艳珍, 崔国庆, 穆志新, 刘龙龙, 李欣, 逯腊虎, 李晓丽, 张晓军. 基于55K SNP芯片的山西冬小麦种质资源遗传多样性分析[J]. 中国农业科学, 2024, 57(10): 1845-1856. |
| [7] | 张一中, 张晓娟, 梁笃, 郭琦, 范昕琦, 聂萌恩, 王绘艳, 赵文博, 杜维俊, 柳青山. 基于表型性状的高粱育种材料遗传多样性分析及综合评价[J]. 中国农业科学, 2023, 56(15): 2837-2853. |
| [8] | 李欢, 鄢小青, 杨占烈, 谭金玉, 黎小冰, 陈能刚, 吴荣菊, 陈惠查, 阮仁超. 贵州香禾糯地方稻种资源表型遗传多样性分析与综合评价[J]. 中国农业科学, 2023, 56(11): 2035-2046. |
| [9] | 姜朋, 张鹏, 姚金保, 吴磊, 何漪, 李畅, 马鸿翔, 张旭. 宁麦系列小麦品种的性状特点及相关基因位点分析[J]. 中国农业科学, 2022, 55(2): 233-247. |
| [10] | 李晓川,王朝海,周平,马维,吴瑞,宋治豪,梅艳. 马铃薯品种(系)田间晚疫病抗性评价和全基因组遗传多样性分析[J]. 中国农业科学, 2022, 55(18): 3484-3500. |
| [11] | 万映伶,朱梦婷,刘爱青,金亦佳,刘燕. 中国观赏芍药表型多样性解析与资源评价[J]. 中国农业科学, 2022, 55(18): 3629-3639. |
| [12] | 胡光明,张琼,韩飞,李大卫,李作洲,汪志,赵婷婷,田华,刘小莉,钟彩虹. 猕猴桃属植物通用型SSR分子标记引物的筛选及应用[J]. 中国农业科学, 2022, 55(17): 3411-3425. |
| [13] | 王璐伟,沈志军,李贺欢,潘磊,牛良,崔国朝,曾文芳,王志强,鲁振华. 基于SSR荧光标记的79份桃种质遗传多样性分析[J]. 中国农业科学, 2022, 55(15): 3002-3017. |
| [14] | 陈旭,郝雅琼,聂兴华,杨海莹,刘松,王雪峰,曹庆芹,秦岭,邢宇. 板栗总苞和坚果主要性状与SSR标记的关联分析[J]. 中国农业科学, 2022, 55(13): 2613-2628. |
| [15] | 徐晓,任根增,赵欣蕊,常金华,崔江慧. 中国高粱地方品种和育成品种穗部表型性状精准鉴定及综合评价[J]. 中国农业科学, 2022, 55(11): 2092-2108. |
|
||