













中国农业科学 ›› 2025, Vol. 58 ›› Issue (22): 4656-4672.doi: 10.3864/j.issn.0578-1752.2025.22.008
收稿日期:2025-07-29
接受日期:2025-10-18
出版日期:2025-11-16
发布日期:2025-11-21
通信作者:
联系方式:
王凡,E-mail:2320951333@qq.com。
基金资助:
WANG Fan( ), LIU ChenWei, LU HongChen, XU RenChao, BIAN XiaoChun(
), LIU ChenWei, LU HongChen, XU RenChao, BIAN XiaoChun( )
)
Received:2025-07-29
Accepted:2025-10-18
Published:2025-11-16
Online:2025-11-21
摘要:
【目的】叶斑病是限制蚕豆生产的一种新型真菌病害,交链格孢(Alternaria alternata)是其主要病原菌之一。本研究通过转录组分析和抗病基因功能验证,探讨蚕豆响应交链格孢侵染的分子途径。【方法】以蚕豆品种CD-006为试验材料,对接种交链格孢后0、6、12和24 h的蚕豆叶片进行转录组测序,筛选差异表达基因(differentially expressed gene,DEG),并对其进行GO(gene ontology)功能注释和KEGG(Kyoto encyclopedia of genes and genomes)代谢通路富集分析,从而挖掘抗病相关基因。采用qRT-PCR(quantitative real-time PCR)验证转录组测序结果,从中筛选出VfPR4进行烟草的遗传转化,并通过有伤接种法完成VfPR4的抗病功能分析。【结果】侵染后6、12和24 h,蚕豆中分别有3 537、3 152和2 947个差异基因上调表达,分别有1 181、1 453和1 319个差异基因下调表达。GO分析发现差异基因主要富集于氧化还原酶活性和一些生物合成(代谢)过程,KEGG分析发现差异基因主要富集于次生代谢物的生物合成、苯丙烷生物合成、类黄酮生物合成等途径。侵染后6 h,KEGG还富集于MAPK信号途径和植物-病原菌互作途径,这2条抗病通路共同富集了32个差异基因。转录组分析发现52个PR,其中35个基因差异表达。选取6个差异基因进行qRT-PCR验证,验证结果与转录组测序结果一致,其中VfPR4在交链格孢侵染后24 h内持续上调表达。烟草遗传转化结果表明,转基因烟草的病斑症状较轻,病斑面积分别为128.94、110.57和92.92 mm2,明显小于野生型病斑面积(174.32 mm2)。【结论】通过转录组分析,发现蚕豆可能主要通过氧化爆发、次生代谢物生物合成、苯丙烷生物合成、MAPK信号途径和植物-病原菌互作等途径响应交链格孢的侵染。VfPR4能够正向调控烟草对交链格孢的抗性。
王凡, 刘陈玮, 陆红臣, 徐仁超, 卞晓春. 蚕豆响应交链格孢侵染的转录组分析及VfPR4的抗病功能验证[J]. 中国农业科学, 2025, 58(22): 4656-4672.
WANG Fan, LIU ChenWei, LU HongChen, XU RenChao, BIAN XiaoChun. Transcriptome Analysis of Vicia faba Response to Alternaria alternata Infection and Validation of the Disease Resistance Function of VfPR4[J]. Scientia Agricultura Sinica, 2025, 58(22): 4656-4672.
表1
qRT-PCR、基因克隆及载体构建所用引物"
| 基因Gene | 引物序列Primer sequence (5′-3′) | 扩增效率Efficiency (%) |
|---|---|---|
| qRT-PCR | ||
| Vfaba.Tiffany.R1.1g286840 | F: GGTTTGGGGAGTGGGTCG | 106.73 |
| R: TAGAAGCCTCAGAAGCCGTAGC | ||
| Vfaba.Tiffany.R1.3g068200 (VfPR4) | F: CCTTTTGTGGACCCGTTGG | 99.07 |
| R: GTGACCCGCCTGGTAGCC | ||
| Vfaba.Tiffany.R1.4g083240 | F: GTTTTGGATGACCCCACAGG | 102.75 |
| R: CGCCATTGATTATGTTGGTGAT | ||
| Vfaba.Tiffany.R1.1g351200 | F: AGTTGAGAAGATTTCATTTGAGGCT | 99.43 |
| R: ATCAGGATGAGCAATACAGTAACCC | ||
| Vfaba.Tiffany.R1.1g183400 | F: CATTTTGCCACTACAGACTCTAAGG | 90.74 |
| R: TGGAGGAGAAACCGTTGTCAG | ||
| Vfaba.Tiffany.R1.6g134000 | F: CAGCAAAGAAACAGGTTGAAAAG | 98.66 |
| R: CACACTTGTCATCACAGTTACCTCA | ||
| VfELF1A | F: GTGAAGCCCGGTATGCTTGT | 106.45 |
| R: CTTGAGATCCTTGACTGCAACATT | ||
| NtActin | F: CCTGAGGTCCTTTTCCAACCA | 93.24 |
| R: GGATTCCGGCAGCTTCCATT | ||
| 基因克隆Gene cloning | ||
| VfPR4 | F: ATGGAGAGCACACAGAGAAGTTTAAC | |
| R: TTAGTCACCGCAATTAACAAAGACGT | ||
| 载体构建Vector construction | ||
| VfPR4 | F: AGAACACGGGGGACGAGCTCATGGAGAGCACACAGAGAAGTTTAAC | |
| R: ACCATGGTGTCGACTCTAGAGTCACCGCAATTAACAAAGACGTAG | ||
表2
测序数据统计与质量检测"
| 样本 Sample | 原始数据 Raw reads | 过滤后数据 Clean reads | 原始碱基 Raw bases | 过滤后碱基 Clean bases | Q20 (%) | Q30 (%) | GC (%) |
|---|---|---|---|---|---|---|---|
| CK-1 | 44713886 | 44544524 | 6707082900 | 6649756373 | 97.21 | 92.37 | 43.04 |
| CK-2 | 37108856 | 37009168 | 5566328400 | 5528310812 | 97.01 | 92.01 | 43.02 |
| CK-3 | 38092376 | 37994204 | 5713856400 | 5674495947 | 97.32 | 92.52 | 43.08 |
| T6-1 | 41623938 | 41522300 | 6243590700 | 6203365542 | 97.19 | 92.36 | 42.81 |
| T6-2 | 50863996 | 50705276 | 7629599400 | 7545767138 | 98.54 | 95.56 | 43.20 |
| T6-3 | 49319542 | 49134708 | 7397931300 | 7333695696 | 97.51 | 93.00 | 43.00 |
| T12-1 | 44038582 | 43868258 | 6605787300 | 6563061275 | 97.26 | 92.50 | 42.86 |
| T12-2 | 38431570 | 38286054 | 5764735500 | 5723593977 | 97.22 | 92.45 | 43.02 |
| T12-3 | 45024378 | 44864168 | 6753656700 | 6702567067 | 97.27 | 92.43 | 42.88 |
| T24-1 | 45809802 | 45609948 | 6871470300 | 6807440098 | 97.47 | 92.84 | 42.98 |
| T24-2 | 45742880 | 45613266 | 6861432000 | 6807387734 | 98.41 | 95.15 | 43.21 |
| T24-3 | 48451546 | 48287776 | 7267731900 | 7192462021 | 98.61 | 95.66 | 43.24 |
表3
比对参考基因组情况统计"
| 样本 Sample | 过滤后数据 Clean reads | 比对到基因组的数据及占比 Total mapped (%) | 比对到基因组唯一位置的数据及占比 Unique mapped (%) |
|---|---|---|---|
| CK-1 | 44092194 | 39123402 (88.73) | 36596547 (83.00) |
| CK-2 | 36492758 | 32535363 (89.16) | 30511820 (83.61) |
| CK-3 | 37415614 | 33617214 (89.85) | 31573700 (84.39) |
| T6-1 | 40849472 | 36455382 (89.24) | 34177712 (83.67) |
| T6-2 | 49378582 | 43572672 (88.24) | 40509613 (82.04) |
| T6-3 | 48266952 | 42929051 (88.94) | 40007796 (82.89) |
| T12-1 | 43210162 | 38477310 (89.05) | 36178498 (83.73) |
| T12-2 | 37742640 | 33652558 (89.16) | 31555095 (83.61) |
| T12-3 | 44271762 | 39224804 (88.60) | 36739592 (82.99) |
| T24-1 | 44969738 | 39722019 (88.33) | 37244088 (82.82) |
| T24-2 | 45022382 | 39229015 (87.13) | 36940861 (82.05) |
| T24-3 | 47568146 | 41713080 (87.69) | 39012843 (82.01) |
表4
比对参考基因组区域情况统计"
| 样本 Sample | 比对到外显子区域的比例 Proportion of comparison to exon regions (%) | 比对到内含子区域的比例 Proportion of comparison to intron regions (%) | 比对到基因间区域的比例 Proportion of comparison to intergenic regions (%) |
|---|---|---|---|
| CK-1 | 83.46 | 4.44 | 12.10 |
| CK-2 | 83.75 | 4.27 | 11.98 |
| CK-3 | 84.03 | 4.10 | 11.88 |
| T6-1 | 83.30 | 4.50 | 12.20 |
| T6-2 | 83.35 | 4.45 | 12.20 |
| T6-3 | 83.52 | 4.37 | 12.11 |
| T12-1 | 82.88 | 4.63 | 12.49 |
| T12-2 | 83.15 | 4.39 | 12.45 |
| T12-3 | 83.12 | 4.58 | 12.31 |
| T24-1 | 82.19 | 4.76 | 13.05 |
| T24-2 | 82.11 | 4.65 | 13.24 |
| T24-3 | 81.98 | 4.71 | 13.31 |
表5
交链格孢侵染后24 h内持续上调表达的6个抗病相关基因"
| 基因编号 Gene ID | 基因注释 Gene annotation | 表达水平Expression level (FPKM) | |||
|---|---|---|---|---|---|
| 0 h | 6 h | 12 h | 24 h | ||
| Vfaba.Tiffany.R1.3g072280 | RBOHB(MAPK信号途径MAPK signaling pathway、植物-病原菌互作Plant-pathogen interaction) | 0.131 | 0.163 | 0.607 | 1.807 |
| Vfaba.Tiffany.R1.1g350240 | PR1(MAPK信号途径MAPK signaling pathway、植物-病原菌互作Plant-pathogen interaction) | 0.663 | 18.979 | 23.724 | 24.035 |
| Vfaba.Tiffany.R1.1g351200 | PR10 | 2.033 | 140.937 | 252.342 | 426.230 |
| Vfaba.Tiffany.R1.1g351320 | PR10 | 0.077 | 0.850 | 2.324 | 27.403 |
| Vfaba.Tiffany.R1.1g351400 | PR10 | 1.868 | 17.391 | 67.293 | 82.358 |
| Vfaba.Tiffany.R1.3g068200 | PR4 | 1.363 | 48.051 | 253.160 | 530.807 |
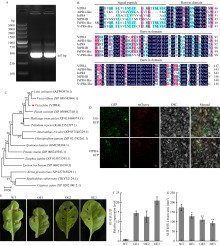
图7
VfPR4增强烟草对交链格孢的抗性 A:VfPR4克隆Cloning of VfPR4;B:VfPR4与其他PR4蛋白的氨基酸序列比对Amino acid sequence alignment of VfPR4 with other PR4 proteins;C:VfPR4蛋白的系统进化树Phylogenetic tree of VfPR4 protein;D:VfPR4的亚细胞定位Subcellular localization of VfPR4;E:接种5 d后的病斑表型Lesion phenotype at 5 d after infection;F:烟草中VfPR4的相对表达量Relative expression level of VfPR4 in tobacco;G:接种5 d后的病斑面积Lesion area at 5 d after infection。*:P<0.05;**:P<0.01"

| [1] |
|
| [2] |
|
| [3] |
李仁慧, 闫智臣, 段廷玉. 蚕豆真菌病害及其研究进展. 草业科学, 2019, 36(8): 1976-1987.
|
|
|
|
| [4] |
|
| [5] |
|
| [6] |
|
| [7] |
|
| [8] |
|
| [9] |
周慧珍, 荣思文, 吴玉珠, 王武, 张嘉, 陈娜, 胡军华. 青脆李果斑病病原鉴定及室内药剂筛选. 中国南方果树, 2023, 52(4): 136-140, 144.
|
|
|
|
| [10] |
|
| [11] |
|
| [12] |
|
| [13] |
|
| [14] |
|
| [15] |
|
| [16] |
|
| [17] |
|
| [18] |
|
| [19] |
|
| [20] |
|
| [21] |
|
| [22] |
卞晓春, 王凡, 刘陈玮, 徐仁超, 陆红臣, 吴春芳. 江苏省蚕豆叶斑病病原菌的分离鉴定、生物学特性与防控药剂筛选. 福建农业学报, 2024, 39(11): 1298-1305.
|
|
|
|
| [23] |
王凡, 卞晓春, 刘陈玮, 徐仁超, 陆红臣, 吴春芳. 南通市蚕豆赤斑病病原菌鉴定及其抑菌药剂筛选. 江苏农业学报, 2024, 40(10): 1810-1817.
|
|
|
|
| [24] |
蔚书涵, 秦小杰, 吴其超, 李玲, 臧德奎, 马燕. 芍药响应细极链格孢侵染的转录组分析. 中国农业科学, 2024, 57(15): 3035-3052. doi: 10.3864/j.issn.0578-1752.2024.15.010.
|
|
|
|
| [25] |
|
| [26] |
|
| [27] |
|
| [28] |
|
| [29] |
|
| [30] |
|
| [31] |
|
| [32] |
|
| [33] |
|
| [34] |
|
| [35] |
|
| [36] |
黄颖. 山新杨响应细链格孢菌侵染的转录组分析及PdbMYB85基因功能的研究[D]. 哈尔滨: 东北林业大学, 2022.
|
|
|
|
| [37] |
李婕婷. 杜鹃花瓣对链格孢菌侵染的转录组和代谢组分析及植物源制剂抑菌活性研究[D]. 贵阳: 贵州师范大学, 2023.
|
|
|
|
| [38] |
|
| [39] |
|
| [40] |
|
| [41] |
|
| [42] |
杨敏. 多组学揭示魔芋响应胡萝卜软腐果胶杆菌侵染的机制研究[D]. 昆明: 云南农业大学, 2023.
|
|
|
|
| [43] |
|
| [44] |
|
| [45] |
|
| [46] |
|
| [47] |
|
| [48] |
|
| [49] |
|
| [50] |
|
| [51] |
|
| [52] |
|
| [53] |
肖胜华, 董贤镘, 彭鑫, 李安子, 闭兆福, 廖铭静, 黄礼豪, 管倩倩, 胡琴, 朱龙付. 转录因子GhWRKY41促进水杨酸合成增强棉花对黄萎病菌的抗性. 作物学报, 2024, 50(10): 2447-2457.
|
|
|
|
| [54] |
|
| [55] |
|
| [56] |
|
| [57] |
|
| [58] |
裴晓兔. 罹病茎瘤芥PR-4蛋白基因的克隆与诱导表达分析[D]. 重庆: 重庆大学, 2013.
|
|
|
|
| [59] |
王自娥, 李有玉, 邓婕, 王瀚林, 孙皓, 陈晓华, 葛锋, 刘迪秋. 岷江百合病程相关蛋白4基因及其启动子的克隆与分析. 植物生理学报, 2022, 58(9): 1747-1756.
|
|
|
|
| [60] |
|
| [1] | 张义茹, 韩雪, 姚鑫杰, 冯军, 魏爱丽, 李文超, 张彬, 韩渊怀, 李红英. 基于多组学解析谷子后熟米色变化的分子机制[J]. 中国农业科学, 2025, 58(9): 1702-1718. |
| [2] | 邹晓威, 夏蕾, 朱晓敏, 孙辉, 周琦, 齐霁, 张亚封, 郑岩, 姜兆远. 基于转录组测序的玉米瘤黑粉菌UM01240过表达菌株诱导玉米抗病性分析[J]. 中国农业科学, 2025, 58(6): 1116-1130. |
| [3] | 孙萍, 朱文灿, 林贤锐, 吴嘉颀, 曹译文, 陈辰斐, 王轶, 朱建锡, 贾惠娟, 钱敏杰, 沈建生. 基于代谢组和转录组解析多雨寡照对桃果皮着色和类黄酮积累的影响[J]. 中国农业科学, 2025, 58(6): 1173-1194. |
| [4] | 谢露露, 李福, 张思远, 高建昌. 基于跨物种转录组解析影响不定根发生的保守基因[J]. 中国农业科学, 2025, 58(6): 1195-1209. |
| [5] | 高岩浩, 王婷婷, 白卫卫, 杜兴杰, 刘贤, 秦本源, 付彤, 孙宇, 高腾云, 张天留. 脂质组与转录组联合揭示南阳牛不同肌肉组织脂质特征的差异表达模式[J]. 中国农业科学, 2025, 58(6): 1239-1258. |
| [6] | 穆赢通, 路景诗, 张雨桐, 石凤翎. 基于转录组和WGCNA的直立型花苜蓿抗旱关键基因识别[J]. 中国农业科学, 2025, 58(21): 4528-4543. |
| [7] | 潘媛, 王德, 刘楠, 孟祥龙, 戴蓬博, 李波, 胡同乐, 王树桐, 曹克强, 王亚南. 两种高通量测序技术鉴定苹果病毒效果评价及两种新病毒的鉴定[J]. 中国农业科学, 2025, 58(2): 266-280. |
| [8] | 林彦, 郑玲玲, 田嘉, 温玥, 陈晨, 王磊. 基于转录组和蛋白质组揭示库尔勒香梨萼片脱落的分子机制[J]. 中国农业科学, 2025, 58(18): 3744-3765. |
| [9] | 董雪, 陈梦秋, 邵晋, 吴学友, 唐培安. 基于WGCNA的稻谷储藏期间差异基因挖掘与品质调控网络构建[J]. 中国农业科学, 2025, 58(14): 2885-2903. |
| [10] | 徐秋韵, 周伟迪, 韩成龙, 谷艳杰. 不同磷肥施用量对高寒地区覆膜蚕豆光合特性、产量和水分利用效率的影响[J]. 中国农业科学, 2025, 58(10): 1917-1933. |
| [11] | 曹言勇, 程泽强, 马娟, 杨文博, 朱卫红, 孙新艳, 李慧敏, 夏来坤, 段灿星. 整合转录组和代谢组学分析揭示玉米对层出镰孢茎腐病的响应机制[J]. 中国农业科学, 2025, 58(1): 75-90. |
| [12] | 戚仁洁, 宁宇, 刘静, 刘之洋, 徐海, 罗志丹, 陈龙正. 基于转录组测序的苦瓜皂苷合成相关基因的鉴定和分析[J]. 中国农业科学, 2024, 57(9): 1779-1793. |
| [13] | 林蔚, 吴水金, 李跃森. 转录组和蛋白质组关联分析解析巴西蕉幼苗响应低温的分子机制[J]. 中国农业科学, 2024, 57(8): 1575-1591. |
| [14] | 高晨曦, 郝陆洋, 胡悦, 李永祥, 张登峰, 李春辉, 宋燕春, 石云素, 王天宇, 黎裕, 刘旭洋. 干旱条件下玉米转座子插入关联的表观调控分析[J]. 中国农业科学, 2024, 57(6): 1034-1048. |
| [15] | 熊尚烨, 张翔, 梁宝慧, 叶仰东, 李浴阳, 朱晓, 朱志鸿, 官华忠, 张帅, 吴建国, 胡杰. 水稻抗褐飞虱基因QBPH1和QBPH4的精细定位与聚合效应分析[J]. 中国农业科学, 2024, 57(23): 4619-4631. |
|
||