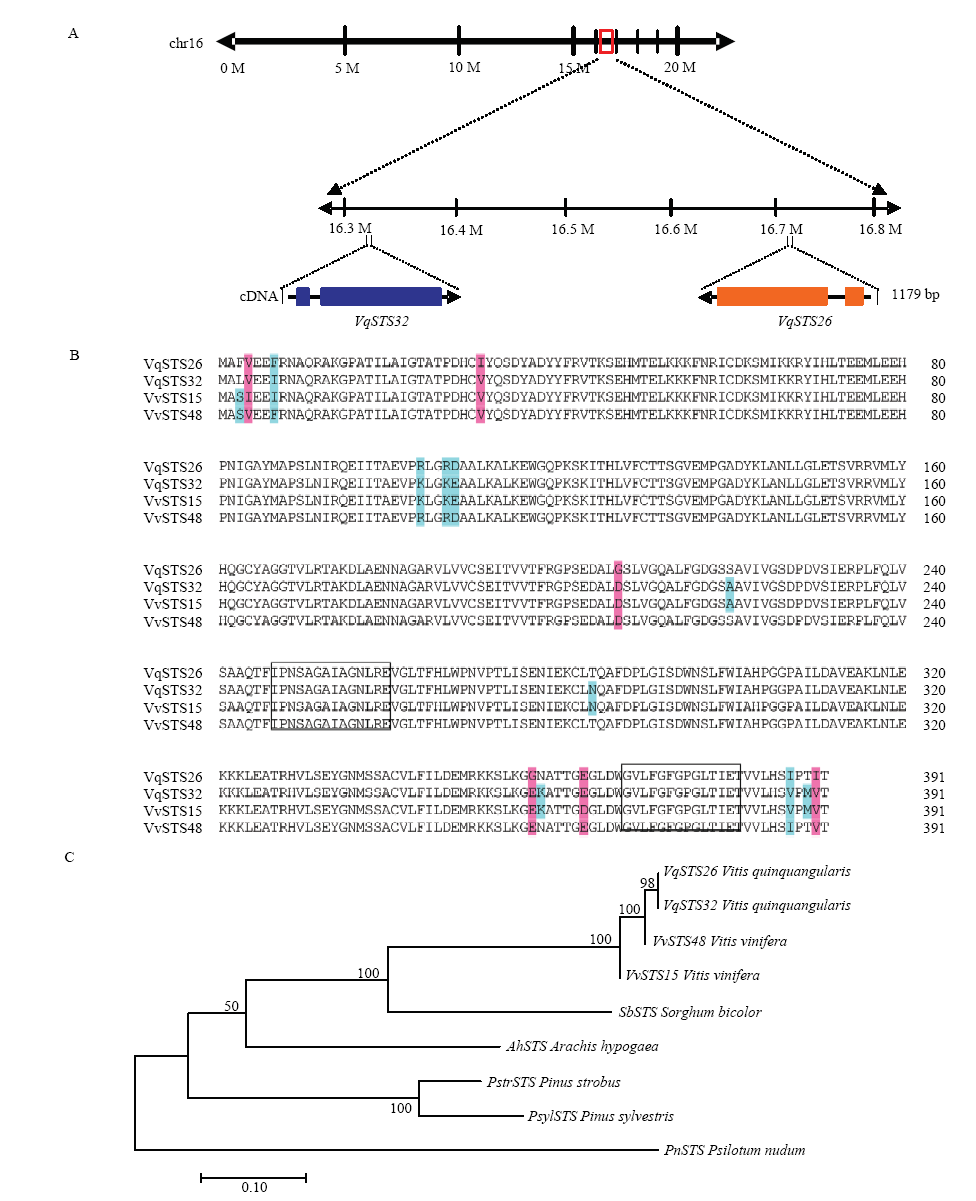
图1. ‘丹凤-2’VqSTS26、VqSTS32的定位、序列比对及聚类分析
A:VqSTS26、VqSTS32的染色体定位分析Chromosome localization analysis of VqSTS26, VqSTS32;B:VqSTS26、VqSTS32与欧洲葡萄同源蛋白氨基酸序列比对;阴影部分为差异位点,方框部分为保守序列Amino acid sequence alignment of homologous proteins between VqSTS26, VqSTS32 and STS genes from V. vinifera, the shading is the difference site and the box part is the conservative sequence;C:VqSTS26、VqSTS32与欧洲葡萄、高粱、花生、白松、樟子松和松叶兰芪合成酶基因氨基酸序列聚类分析,VqSTS26(AFM56643.1)、VqSTS32(AFM56649.1)、VvSTS15(XP_002268756.1)、VvSTS48(NP_001267934.1)、SbSTS(AAL49965)、AhSTS(BAA78617)、PstrSTS(CAA87012)、PsylSTS(CAA43165)、PnSTS(BAA87924)Cluster analysis of STS amino acid sequence from VqSTS26, VqSTS32 and V. vinifera, Sorghum bicolor, Arachis hypogaea, Pinus strobus, Pinus sylvestris, Psilotum nudum
Fig. 1. Localization, sequence alignment and cluster analysis of VqSTS26 and VqSTS32 from ‘Danfeng-2’